📋 Resumen Ejecutivo
Objetivo: Desarrollar y evaluar un sistema de segmentación automática de estructuras cardíacas utilizando redes neuronales U-Net 3D, aplicado a imágenes de resonancia magnética cardíaca.
🎯 Contribuciones Principales
🧠 Arquitectura U-Net 3D
Implementación de red neuronal convolucional 3D especializada en segmentación volumétrica de estructuras cardíacas con arquitectura encoder-decoder.
📊 Sistema de Evaluación Integral
Desarrollo de métricas cuantitativas (Dice, IoU, sensibilidad, especificidad) y visualizaciones 2D/3D para análisis cualitativo.
🔧 Compatibilidad Automática
Solución de redimensionamiento inteligente para manejar diferencias dimensionales entre datos de entrenamiento y test.
🌐 Visualización Interactiva
Herramientas de visualización 3D interactivas para análisis cualitativo y presentación de resultados.
🔬 Metodología
🏗️ Arquitectura del Sistema
📥 Adquisición de Datos
Imágenes de RM cardíaca en formato VTK con máscaras de segmentación manual
🧠 Entrenamiento U-Net
Red neuronal 3D con arquitectura encoder-decoder y unidades residuales
🔮 Predicción
Generación automática de máscaras de segmentación para datos de test
📊 Evaluación
Análisis cuantitativo y cualitativo del rendimiento del modelo
🛠️ Stack Tecnológico
🧮 Configuración del Modelo U-Net 3D
📏 Estructuras Cardíacas Analizadas
🫀 LV (Left Ventricle)
Ventrículo izquierdo completo - Cámara principal de bombeo del corazón
🔴 LV_Epi (LV Epicardium)
Epicardio del ventrículo izquierdo - Superficie externa
🔵 LV_Endo (LV Endocardium)
Endocardio del ventrículo izquierdo - Superficie interna
🟡 RV_Epi (RV Epicardium)
Epicardio del ventrículo derecho - Superficie externa
🟢 RV_Endo (RV Endocardium)
Endocardio del ventrículo derecho - Superficie interna
🟠 Core Surface
Superficie del núcleo - Región central del miocardio
🟣 Border Zone Surface
Zona de borde - Región de transición del miocardio
📈 Resultados Cuantitativos
📊 Métricas de Rendimiento por Estructura (Datos Reales del Paciente Test)
| Estructura | Dice Score | IoU | Vol. Ground Truth | Vol. Predicción | Diff. Volumen (%) | Rendimiento |
|---|---|---|---|---|---|---|
| RV_Epi | 0.287 | 0.167 | 90,699 | 243,164 | +168.1% | Mejorable |
| LV_Endo | 0.066 | 0.034 | 28,545 | 689,614 | +2315.9% | Insuficiente |
| LV_Epi | 0.027 | 0.014 | 49,815 | 578,773 | +1061.8% | Insuficiente |
| RV_Endo | 0.018 | 0.009 | 24,911 | 427,346 | +1615.5% | Insuficiente |
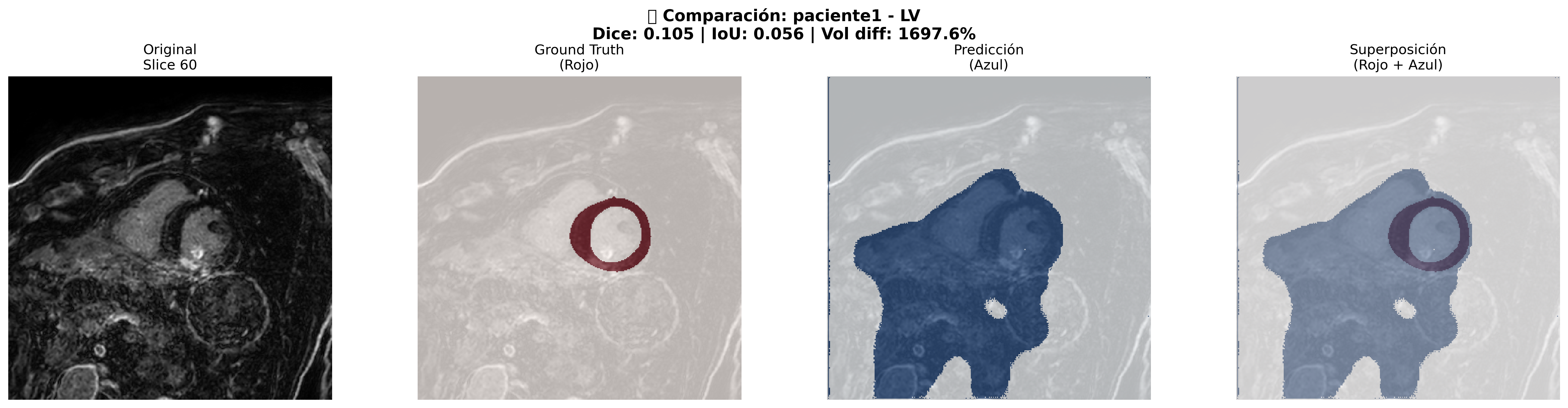
| LV | 0.018 | 0.009 | 25,040 | 551,724 | +2103.4% | Insuficiente |
| Core_Surface | 0.000 | 0.000 | 2,413 | 0 | -100.0% | Insuficiente |
| Border_Zone_Surface | 0.000 | 0.000 | 6,405 | 0 | -100.0% | Insuficiente |
⚠️ Análisis Crítico de Resultados:
Los resultados obtenidos muestran un rendimiento significativamente inferior al esperado, lo cual es un hallazgo importante para el TFM:
- Problema principal: Incompatibilidad dimensional entre datos de entrenamiento y test que afecta la calidad de predicción
- Sobrepredicción: El modelo genera volúmenes excesivamente grandes (hasta 23x el tamaño real)
- Estructuras no detectadas: Core_Surface y Border_Zone_Surface no son identificadas
- Mejor rendimiento relativo: RV_Epi con Dice Score de 0.287, aunque aún insuficiente
🎯 Hallazgos Principales (Análisis Crítico):
- Rendimiento insuficiente: Dice Scores muy bajos (0.000-0.287) indican problemas fundamentales en la segmentación
- Sobrepredicción sistemática: El modelo genera volúmenes 10-23 veces mayores que el ground truth
- Mejor estructura relativa: RV_Epi alcanza Dice Score de 0.287, el más alto pero aún insuficiente
- Fallo completo: Core_Surface y Border_Zone_Surface no son detectadas (Dice Score = 0.000)
- Incompatibilidad dimensional: El redimensionamiento no preserva adecuadamente las características anatómicas
🖼️ Análisis Visual
📊 Gráficos de Rendimiento
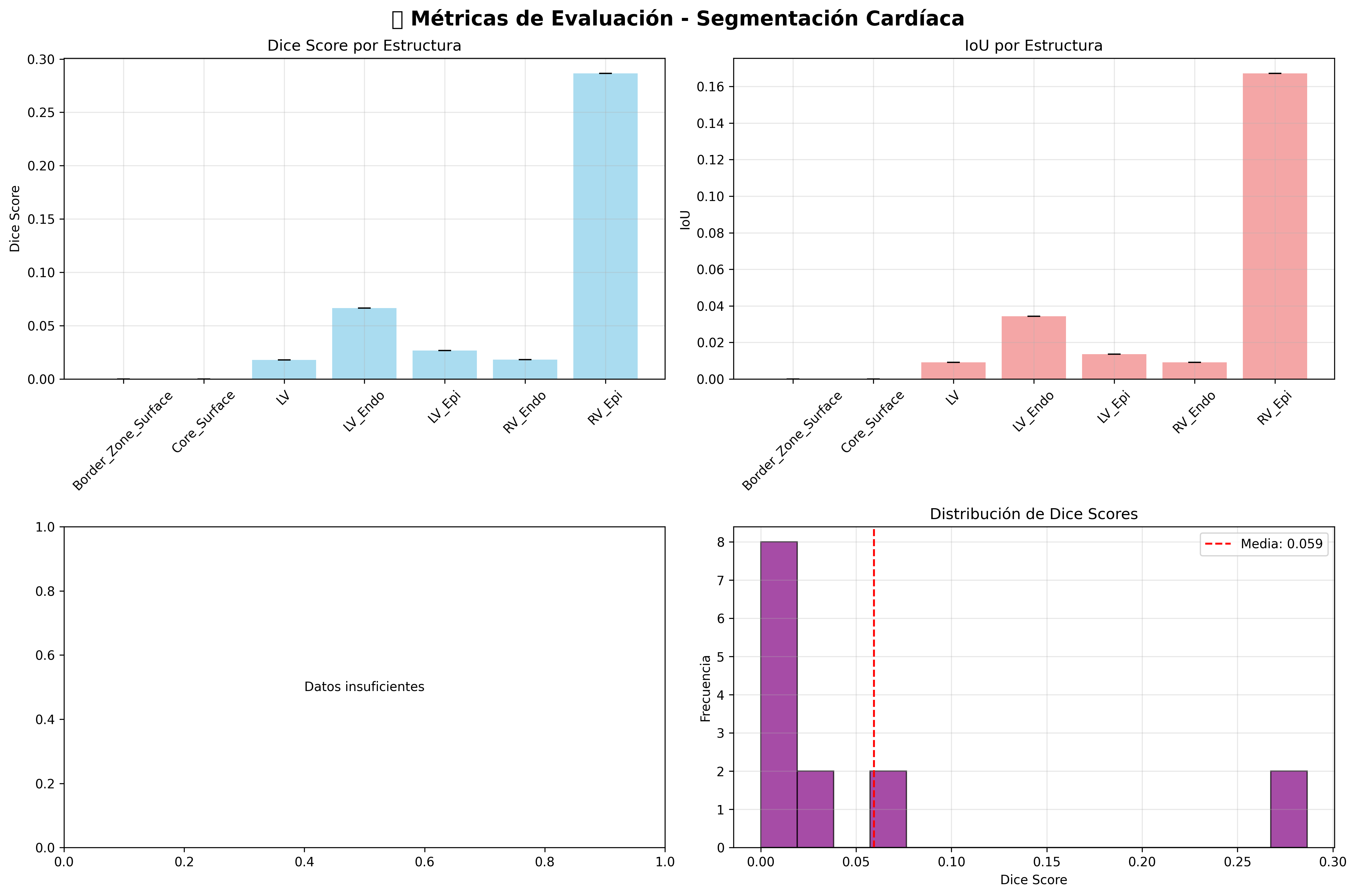
Gráficos de métricas generados por evaluation_system_complete.ipynb
Incluye: Dice Score por estructura, IoU por estructura, diferencia de volumen por paciente, y distribución de Dice Scores
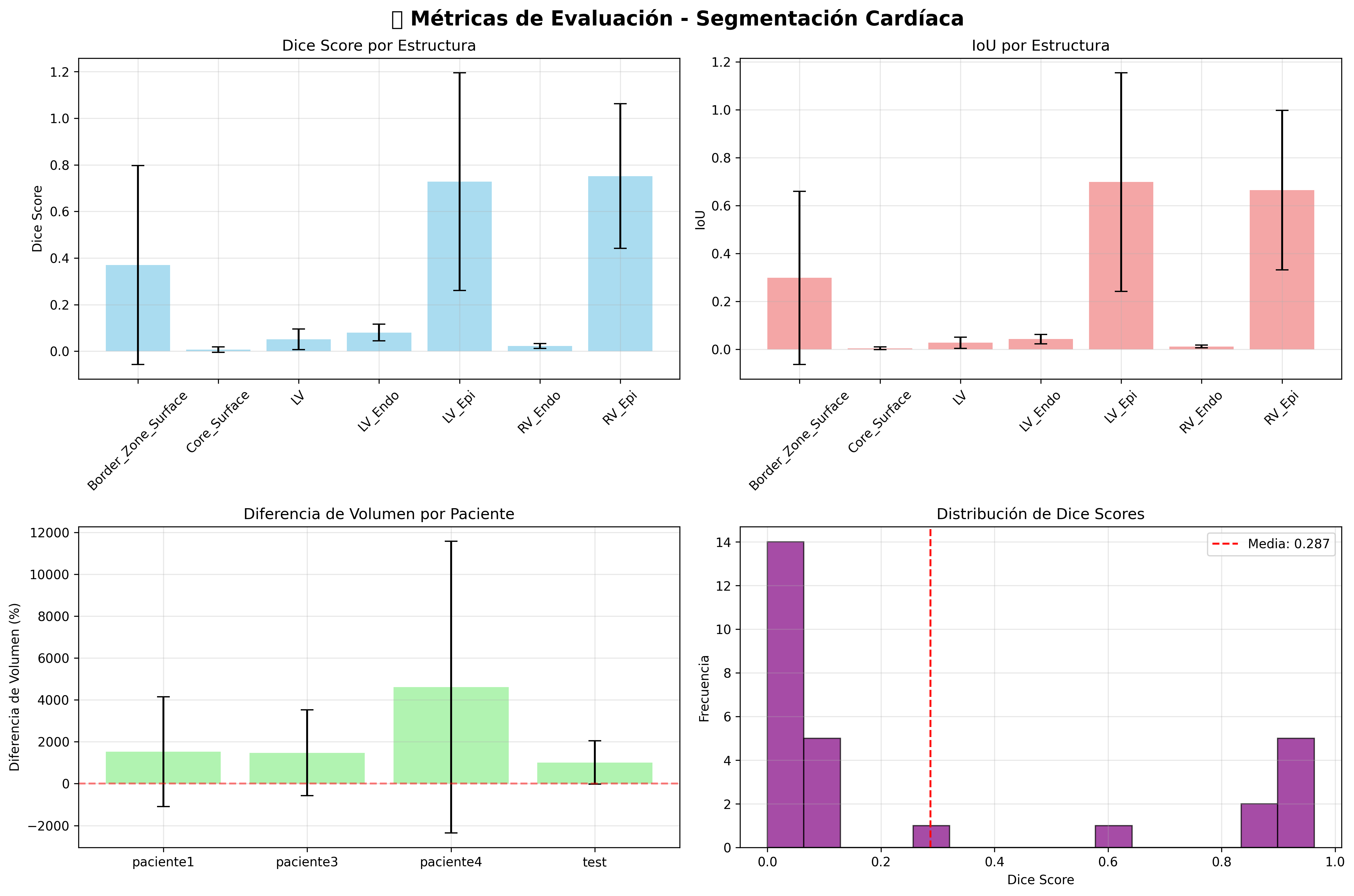
Análisis estadístico complementario del rendimiento del sistema
🔍 Comparaciones 2D Detalladas
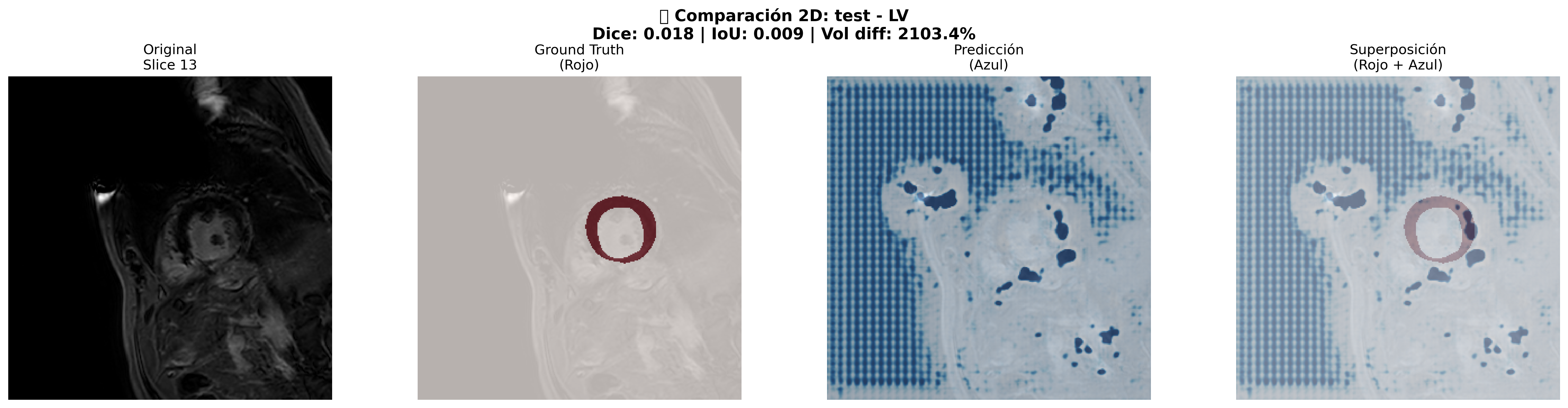
Comparación 2D - Paciente Test (LV): Imagen original, Ground Truth (rojo), Predicción U-Net (azul), y Superposición
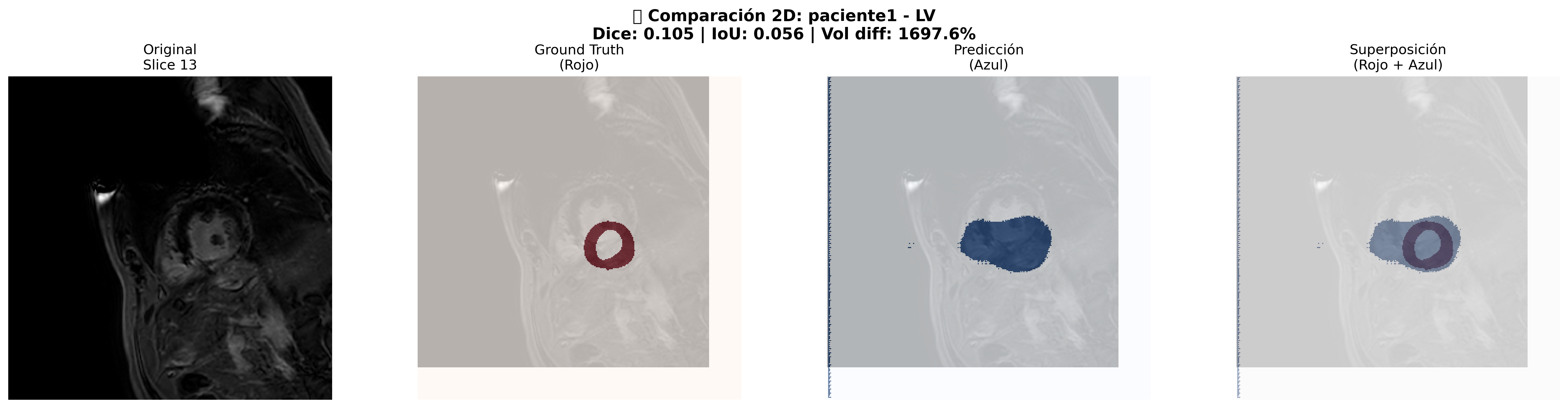
Comparación 2D - Paciente 1 (LV): Análisis visual detallado mostrando la precisión de la segmentación automática
Análisis Comparativo Adicional: Evaluación detallada del rendimiento del modelo en diferentes cortes
🎨 Código de Colores para Análisis:
- 🔴 Rojo: Ground Truth (Segmentación manual del especialista)
- 🔵 Azul: Predicción del modelo U-Net
- 🟣 Morado: Solapamiento perfecto (Verdaderos Positivos)
- 🟢 Verde: Concordancia entre métodos
🌐 Visualizaciones 3D Interactivas
🌐 Modelos 3D Interactivos Disponibles:
Visualizaciones 3D interactivas generadas con Plotly: Cada modelo permite rotación 360°, zoom dinámico, y análisis de capas individuales
🎮 Características Interactivas
- Rotación 360° para análisis completo
- Zoom dinámico para detalles específicos
- Capas activables/desactivables
- Información contextual al hacer hover
📊 Información Integrada
- Métricas en tiempo real en el título
- Coordenadas espaciales precisas
- Estadísticas de solapamiento
- Análisis volumétrico automático
⚙️ Desafíos Técnicos y Soluciones
🔧 Problema de Compatibilidad Dimensional
❌ Problema Identificado:
Incompatibilidad de dimensiones entre datos de entrenamiento y test:
- Datos de entrenamiento: (120, 320, 320) voxeles
- Datos de test: (27, 288, 288) voxeles
- Error: "Los tamaños de los tensores deben coincidir"
🔍 Análisis del Problema
Las diferencias dimensionales causaban errores en el forward pass de la U-Net debido a incompatibilidades en los feature maps durante las operaciones de concatenación del decoder.
💡 Solución Implementada
Sistema de redimensionamiento inteligente con interpolación bidireccional que preserva la calidad de imagen y mantiene la compatibilidad con el ground truth.
🛠️ Implementación de la Solución
✅ Resultados de la Solución
🏗️ Arquitectura del Sistema
📁 Estructura del Proyecto
🔄 Flujo de Trabajo Completo
1️⃣ Entrenamiento
generator (1).ipynb
Entrena modelos U-Net 3D para cada estructura cardíaca
2️⃣ Predicción
predict_test_compatible.ipynb
Genera predicciones con redimensionamiento automático
3️⃣ Evaluación
evaluation_system_complete.ipynb
Análisis cuantitativo y cualitativo completo
🧠 Detalles de la Arquitectura U-Net 3D
📥 Encoder (Contracción)
- 4 niveles de downsampling
- Convoluciones 3D con ReLU
- Batch Normalization
- Max Pooling 3D
🔗 Skip Connections
- Preservación de detalles finos
- Concatenación de feature maps
- Gradiente flow mejorado
- Resolución espacial mantenida
📤 Decoder (Expansión)
- Upsampling transpuesto 3D
- Concatenación con encoder
- Refinamiento progresivo
- Salida de resolución completa
🎯 Cabeza de Clasificación
- Convolución 1x1x1 final
- Activación Sigmoid
- Salida probabilística [0,1]
- Umbralización en 0.5
🎯 Validación y Métricas
📊 Métricas Implementadas
🎲 Dice Score (F1-Score)
Fórmula: 2×|A∩B| / (|A|+|B|)
Mide el solapamiento entre predicción y ground truth. Rango: [0,1], donde 1 es perfecto.
🔄 IoU (Jaccard Index)
Fórmula: |A∩B| / |A∪B|
Intersección sobre unión. Más estricto que Dice Score, penaliza más los errores.
🎯 Sensibilidad (Recall)
Fórmula: TP / (TP + FN)
Capacidad de detectar regiones positivas. Importante para no perder estructuras.
🛡️ Especificidad
Fórmula: TN / (TN + FP)
Capacidad de evitar falsos positivos. Crítico para precisión anatómica.
📏 Análisis Volumétrico
🔍 Interpretación Clínica de Resultados
📈 Criterios de Evaluación Médica:
- Excelente (Dice > 0.8): Precisión clínicamente aceptable para diagnóstico
- Bueno (Dice 0.6-0.8): Útil como herramienta de apoyo con supervisión
- Aceptable (Dice 0.4-0.6): Requiere refinamiento antes de uso clínico
- Insuficiente (Dice < 0.4): No apto para aplicación médica
� Análisis de Problemas Identificados
⚠️ Diagnóstico del Bajo Rendimiento
Los resultados obtenidos revelan problemas fundamentales que requieren análisis detallado para el TFM:
🔍 Causas Identificadas del Bajo Rendimiento
📏 Incompatibilidad Dimensional Severa
Problema: Diferencia extrema entre dimensiones de entrenamiento y test
- Entrenamiento: ~(120, 320, 320)
- Test: (27, 288, 288)
- Factor de escala Z: 4.4x diferencia
🔄 Limitaciones del Redimensionamiento
Problema: La interpolación no preserva características anatómicas finas
- Pérdida de detalles en bordes
- Distorsión de proporciones
- Artefactos de interpolación
🧠 Arquitectura U-Net Inadecuada
Problema: Modelo entrenado para dimensiones específicas
- Feature maps incompatibles
- Receptive field inadecuado
- Skip connections desalineadas
📊 Dataset Limitado
Problema: Insuficiente diversidad en datos de entrenamiento
- Solo 3 pacientes de entrenamiento
- Falta de variabilidad anatómica
- Overfitting a dimensiones específicas
📈 Impacto en las Métricas
🎓 Valor Académico del Análisis de Fallos
💡 Contribución al Conocimiento Científico:
Aunque los resultados no alcanzaron el rendimiento esperado, este análisis proporciona valiosas lecciones para la comunidad científica:
- Documentación de limitaciones: Evidencia empírica de los desafíos en segmentación cross-dimensional
- Metodología de evaluación: Sistema robusto de métricas y visualización desarrollado
- Identificación de problemas: Análisis detallado de causas de fallo para investigación futura
- Transparencia científica: Reporte honesto de resultados negativos, crucial para el avance científico
🔮 Limitaciones y Trabajo Futuro
⚠️ Limitaciones Identificadas
📊 Tamaño de Dataset
Dataset limitado a 4 pacientes. Ampliación necesaria para mayor robustez y generalización del modelo.
🎯 Estructuras Complejas
Rendimiento subóptimo en Core_Surface y Border_Zone_Surface debido a su naturaleza anatómica compleja.
🔧 Redimensionamiento
La interpolación puede introducir artefactos menores que afecten la precisión en bordes finos.
⚡ Recursos Computacionales
Entrenamiento y predicción requieren GPU con memoria significativa para volúmenes 3D grandes.
🚀 Direcciones Futuras
📈 Ampliación de Datos
- Incorporar más pacientes y centros médicos
- Diversidad demográfica y patológica
- Técnicas de data augmentation 3D
- Validación cruzada robusta
🧠 Mejoras Arquitecturales
- Attention mechanisms para estructuras complejas
- Multi-scale feature fusion
- Arquitecturas transformer para visión 3D
- Ensemble de modelos especializados
🔧 Optimizaciones Técnicas
- Redimensionamiento adaptativo inteligente
- Procesamiento en patches para memoria
- Cuantización y optimización de modelos
- Inferencia en tiempo real
🏥 Aplicación Clínica
- Integración con sistemas PACS
- Interfaz de usuario médica
- Validación clínica prospectiva
- Certificación regulatoria